在做机器学习的时候,经常会遇到三个特征以上的数据,这类数据通常被称为高维数据。数据做好类别分类后,通过二维图或者三维图进行可视化,对于高维数据可以通过PCA(Principal Component Analysis),即主成分分析方法,是一种使用最广泛的数据降维算法。
PCA的主要思想是将n维特征映射到k维上,这k维是全新的正交特征也被称为主成分,是在原有n维特征的基础上重新构造出来的k维特征。详情可参考降维——PCA。
代码实现
这里以KNN为例,可视化KNN分类高维数据的结果,代码如下:
python">import pandas as pd
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
from sklearn.decomposition import KernelPCA
# 原始数据
path = 'data.csv'
raw_data = pd.read_csv(path, header=0).to_numpy().tolist()
print('原始数据维度:', len(raw_data[0][1:]))
# 坐标归一化,统一尺度
tmp_data = []
for points in raw_data:
points_x, points_y = points[1::2], points[2::2]
max_x, min_x, max_y, min_y = max(points_x), min(points_x), max(points_y), min(points_y)
tmp_data.append(points[:1]) # 存储标签
for x, y in zip(points_x, points_y):
tmp_data[-1].extend([(x - min_x) / (max_x - min_x), (y - min_y) / (max_y - min_y)]) # 存储坐标
raw_data = tmp_data
# PCA降维
pca_data = [i[1:] for i in raw_data]
pca = KernelPCA(n_components=3, kernel='cosine')
pca_data = pca.fit_transform(pca_data).tolist()
print('降维数据维度:', len(pca_data[0]))
# knn预测,保留一个数据作为预测数据,剩下的作为训练数据
k = 5
error = 0
color_cate = {1: 'b', 2: 'r', 3: 'g', 4: 'y'}
plt.figure(figsize=(10, 6))
for index1, item1 in enumerate(zip(raw_data, pca_data)):
raw_points, pca_points = item1[0], item1[1]
x_train, y_train = [], []
for index2, item2 in enumerate(raw_data):
if index1 != index2:
x_train.append(item2[1:])
y_train.append(item2[0])
knn = KNeighborsClassifier(n_neighbors=k, weights='distance')
knn.fit(x_train, y_train)
x_test = [raw_points[1:]]
predict = knn.predict(x_test)
if predict[0] != raw_points[0]:
error += 1
print('预测:', predict[0], '真实:', raw_points[:1])
# 用PCA降维的数据可视化
color = color_cate[predict[0]]
if len(pca_points) == 3:
# 画二维图
plt.subplot(121)
plt.scatter(pca_points[0], pca_points[1], linewidths=0, color=color)
# 画三维图
ax = plt.subplot(122, projection='3d')
ax.scatter(pca_points[0], pca_points[1], pca_points[2], linewidths=0, color=color)
print('错误:', error)
plt.show()
数据格式
我的数据格式如下,第一列为标签,后面为坐标,一个坐标(x,y)为一个特征。

归一化
由于我的数据特征是不同图像的坐标点,尺度不一样,需要对其进行归一化处理。归一化公式:
python"># 坐标归一化,统一尺度
tmp_data = []
for points in raw_data:
points_x, points_y = points[1::2], points[2::2]
max_x, min_x, max_y, min_y = max(points_x), min(points_x), max(points_y), min(points_y)
tmp_data.append(points[:1]) # 存储标签
for x, y in zip(points_x, points_y):
tmp_data[-1].extend([(x - min_x) / (max_x - min_x), (y - min_y) / (max_y - min_y)]) # 存储坐标
raw_data = tmp_data
PCA降维
python"># PCA降维
pca_data = [i[1:] for i in raw_data]
pca = KernelPCA(n_components=3, kernel='cosine')
pca_data = pca.fit_transform(pca_data).tolist()
print('降维数据维度:', len(pca_data[0]))这里用的是核主成分分析方法,个人觉得KernelPCA的数据分布更容易区分。超参数n_components为正整数时,指保留主成分的维数;为 (0,1] 范围的实数时,表示主成分的方差和所占的最小阈值。详情参考:Sklearn主成分分析。
KNN预测
python"># knn预测原始数据,保留一个数据作为预测数据,剩下的作为训练数据
k = 5
error = 0
color_cate = {1: 'b', 2: 'r', 3: 'g', 4: 'y'}
plt.figure(figsize=(10, 6))
for index1, item1 in enumerate(zip(raw_data, pca_data)):
raw_points, pca_points = item1[0], item1[1]
x_train, y_train = [], []
for index2, item2 in enumerate(raw_data):
if index1 != index2:
x_train.append(item2[1:])
y_train.append(item2[0])
knn = KNeighborsClassifier(n_neighbors=k, weights='distance')
knn.fit(x_train, y_train)
x_test = [raw_points[1:]]
predict = knn.predict(x_test)
if predict[0] != raw_points[0]:
error += 1
print('预测:', predict[0], '真实:', raw_points[:1])这里以KNN预测为例,预测原始数据,保留一个数据作为预测数据,剩下的作为训练数据,遍历预测。KNN中的k值指最近邻数据的个数。k个数据中类别数最多的类别即为预测数据的类别,当类别数一致时,可以设置超参数weights为distance,表示不同距离的点有不同的权重,权重更大的即为预测类别。详情参考:K最近邻算法(KNN)。
可视化
python"># 用PCA降维的数据可视化
color = color_cate[predict[0]]
if len(pca_points) == 3:
# 画二维图
plt.subplot(121)
plt.scatter(pca_points[0], pca_points[1], linewidths=0, color=color)
# 画三维图
ax = plt.subplot(122, projection='3d')
ax.scatter(pca_points[0], pca_points[1], pca_points[2], linewidths=0, color=color)当使用PCA降维到3维时,可以同时可视化二维图和三维图。注意:用matplotlib画图读取的数据不能为字符串,否则显示出来会有误。效果如下:
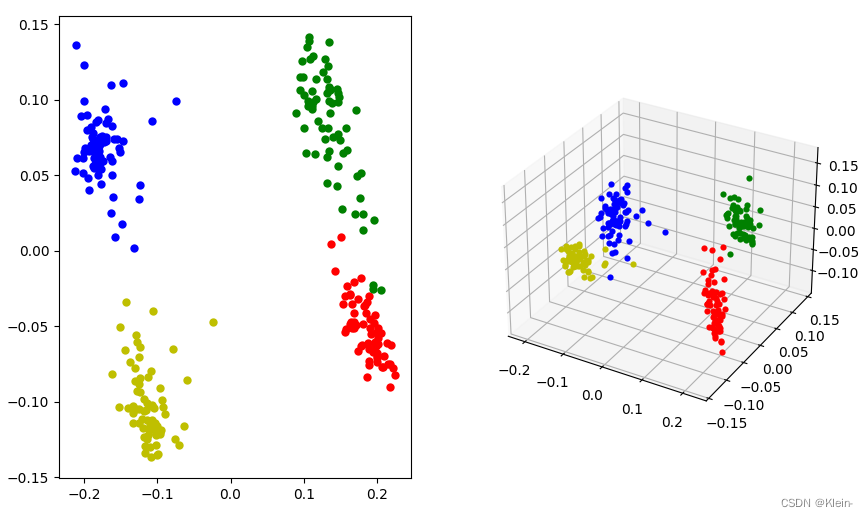
参考链接
【机器学习】降维——PCA(非常详细) - 知乎
Python数模笔记-Sklearn(3)主成分分析 - youcans - 博客园
K最近邻算法(KNN)---sklearn+python实现_zcc_TPJH的博客-CSDN博客_knn python sklearn





